利用荧光标记方法通过DNA点阵成像获取碱基信号,是高通量测序技术的关键步骤之一。华大智造近期重磅发布的CoolMPS高通量测序试剂套装却颠覆过往,开创性地推出了基于抗体的测序试剂产品,它使用的dNTPs 未在碱基上标记荧光。这一创新,可以实现无损碱基识别,能够让碱基识别更为清晰。这一测序试剂套装也即将于圣诞节正式开售。
那么,它的诞生将带来哪些独具一格优势? 它的数据表现如何?
一、优势特点:信号强、天然、效率高
信号强、天然及效率高可以概括地反映CoolMPS高通量测序试剂盒的特点及优势:
首先,它信号强,每个抗体能标记多个荧光,碱基信号更强。每个抗体分子可以标记上多个荧光分子,相同拷贝数的DNB,用CoolMPS可以获得更高的信号和更高的信噪比,从而使测序更准确。图1显示了在多个循环中使用两种测序化学的强度比较,CoolMPS产品信号强度远高于传统高通量测序试剂产品。
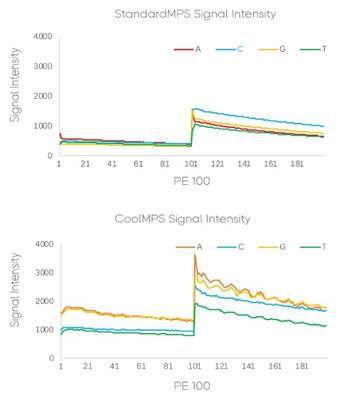
图1 测序时CoolMPS的信号强度及随循环数的变化趋势
其次,它没有非天然碱基带来的荧光淬灭,因为荧光分子标记在抗体上,测序过程中无多余化学键的残留。在传统的高通量测序方法中,聚合的碱基上会留下多余的化学键,即“疤痕”(图2),这可能会影响待测碱基上的荧光信号,进而影响测序的准确性。CoolMPS方法中新合成链的碱基为天然碱基,不会因为碱基上的疤痕而影响荧光信号,最终获得更高的准确性和更长的测序读长。
图3展示了CoolMPS测序中,前两个碱基对被测碱基信号的影响。碱基上不存在任何疤痕,不论前面两个碱基是什么,其A、C、G、T的信号均相当稳定。
图4展示了CoolMPS的测序错误类型与前一个碱基的关系:无论前一个碱基是什么,CoolMPS的测序错误都非常低,且没有明显的错误偏好。
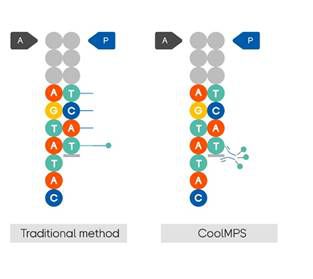
图2 CoolMPS与传统高通量测序方法的比较:无疤痕
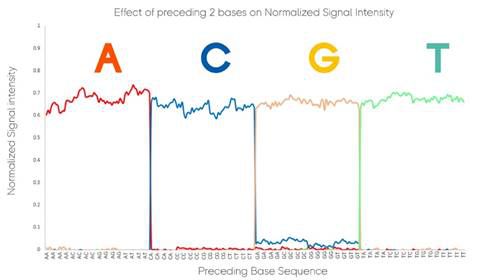
图3 CoolMPS测序中,前两个碱基对被测碱基信号的影响
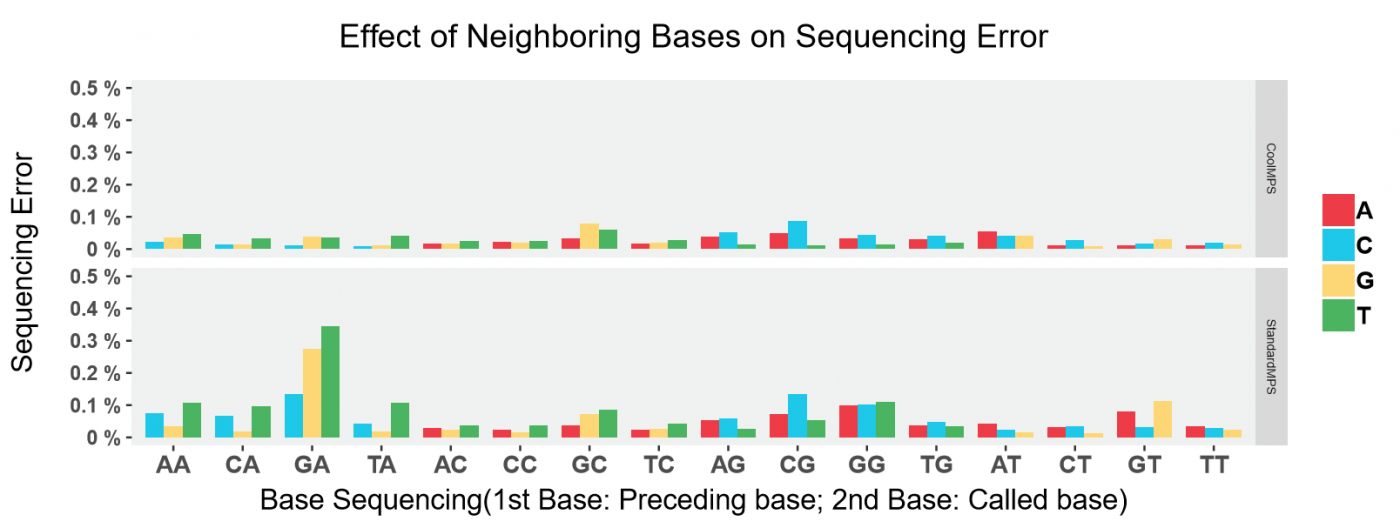
图4 CoolMPS与StandardMPS的比较:前两个碱基对被测碱基信号的影响
再者,它的反应更完全,即DNA聚合天然碱基的效率更高。聚合酶结合及新加的都是天然碱基, DNA聚合酶在生物体内的底物是天然dNTPs。 虽然经过突变体的改造,DNA聚合酶也能加碱基上有庞大荧光基团标记的dNTPs,但其反应效率远远不如加碱基上没有标记的dNTPs。此外每一轮反应接近100%的完成率也是进行长读长测序的必要条件,否则信号会呈现指数下降,产生测序错误。
测序时,常用lag及run on指标来反映反应是否完全。lag是指测序进行到N位置时,其某些拷贝才反应到N-1位置的比例;而runon则指某些拷贝已反应到N+1位置上的比例。我们测定了用于CoolMPS的测序酶在加100%的有荧光标记碱基的dNTPs与加无修饰碱基的cold dNTPs的lag与runon,其结果如图5所示,加cold dNTPs的平均lag值(0.2%)比加荧光标记dNTPs的平均lag值(0.7%)低很多。同时,加cold dNTPs的平均runon值(0.06%)比加荧光标记dNTPs的平均Runon值(0.24%)低很多。CoolMPS技术的低lag及低runon可能使得测序读长更长。
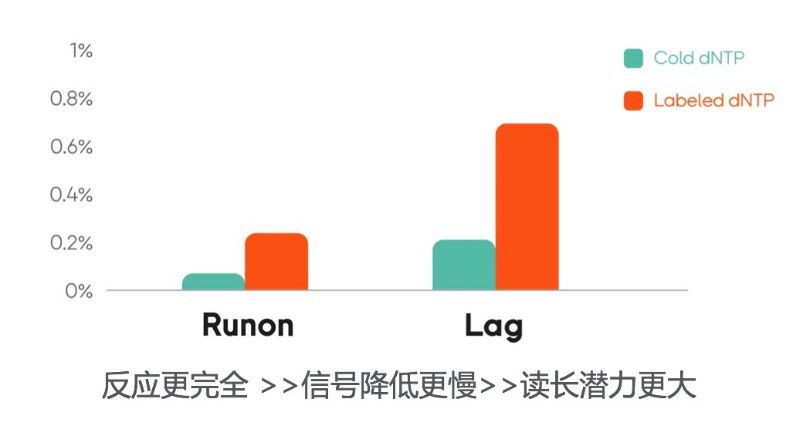
图5 DNA聚合酶对Cold dNTP与Labeled dNTPs的聚合的lag和runon的效率比较
应用数据展示
1. CoolMPS PE100测序数据产量及Q30表现优异
CoolMPS与StandardMPS的PE100测序在WGS(PCR)、WGS(PCR-Free)、WES、RNA-Seq和Panel等应用上的数据显示:CoolMPS的数据产量比StandardMPS高6%,且Q30%比StandardMPS高1~9%。
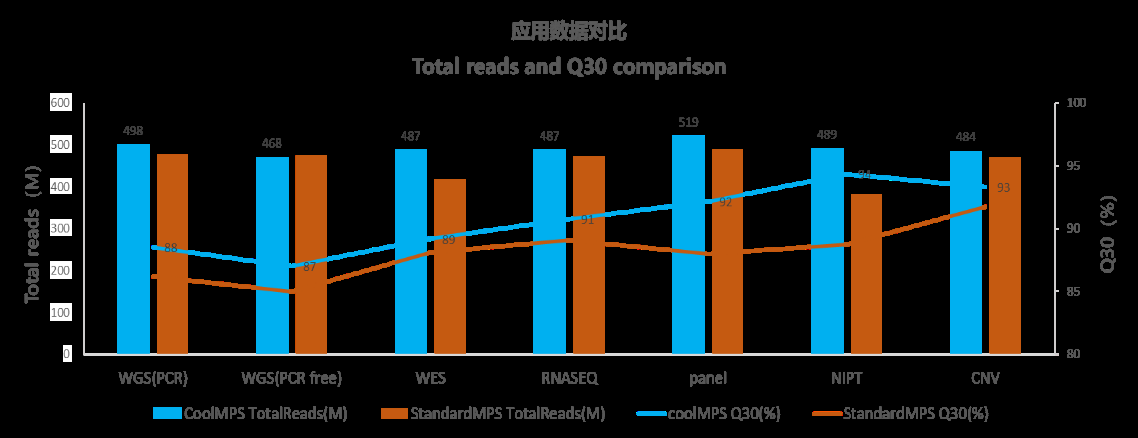
图7 CoolMPS PE100数据产量和数据质量表现(Demo数据)
2. 应用数据表现:外显子、肿瘤Panel、RNA-Seq
针对三种不同的应用数据,结果显示CoolMPS测序数据的clean rate和mapping rate表现良好,且覆盖度均一;CoolMPS检测频率准确,且与理论频率的相关性更高。
-外显子
样本:NA12878
建库:MGIEasy 外显子捕获V5探针试剂套装
测序平台:MGISEQ-2000(DNBSEQ-G400)
测序读长:PE100(CoolMPS PE100试剂)
测序结果:
对4个NA12878样本的WES数据截取100X平均深度的数据,采用MegaBolt软件进行了同步分析比较,如表2和图10所示,CoolMPS测序数据的clean rate和mapping rate表现良好,且覆盖度均一。
表2 CoolMPS的WES分析结果
|
Sample |
WES-NA12878 |
|
Insert-size |
258bp |
|
Average depth |
102X |
|
Rate-clean |
98.22% |
|
Q20-clean |
96.24% |
|
Q30-clean |
88.31% |
|
Mapping Rate |
98.55% |
|
Duplication Rate |
12.24% |
|
Capture Rate on Reads |
63.75% |
|
Uniformity(>0.2f) |
94.51% |
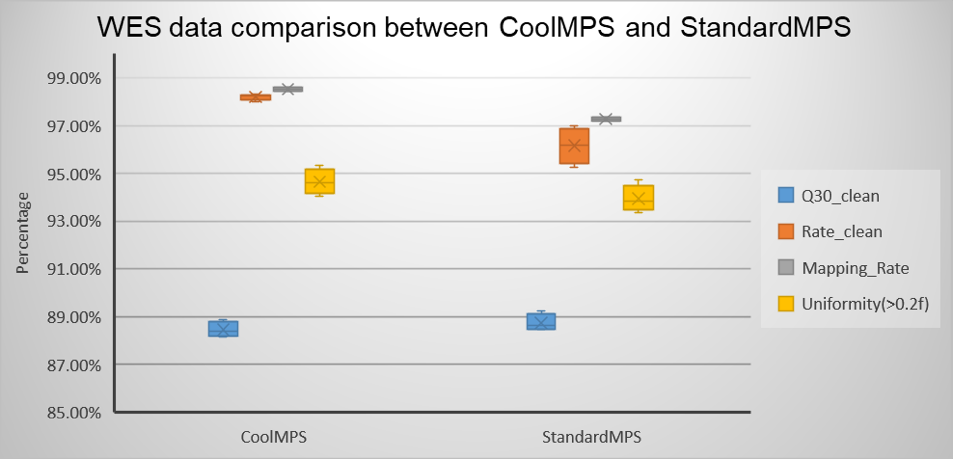
图8 CoolMPS与StandardMPS的WES数据比较
-肿瘤:
样本:FFPE标准品Horizon HD200
测序平台:MGISEQ-2000(DNBSEQ-G400)
测序读长:PE100
测序试剂:CoolMPS & StandardMPS
测序结果:
CoolMPS与StandardMPS检测频率均准确,CoolMPS检测与理论频率的相关性更高。
表3 CoolMPS与StandardMPS的肿瘤数据比较
|
Metrics |
CoolMPS |
StandardMPS |
|
Clean Q30 |
92.1% |
87.2% |
|
Mapping rate |
99.88% |
99.95% |
|
Capture rate |
97.1% |
97.3% |
|
Coverage(>=100X) |
99.4% |
99.7% |
|
Uniformity(>0.2x) |
97.2% |
95.4% |
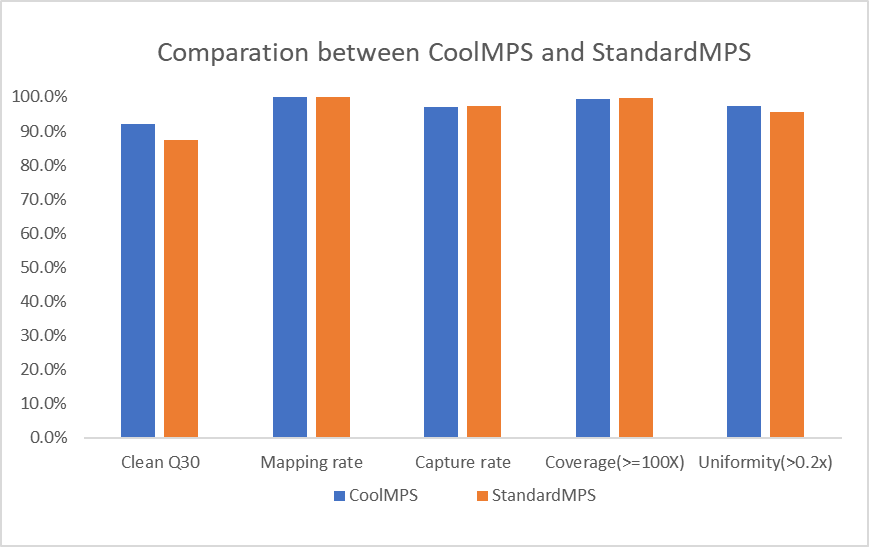
图9 CoolMPS与StandardMPS的肿瘤数据比较
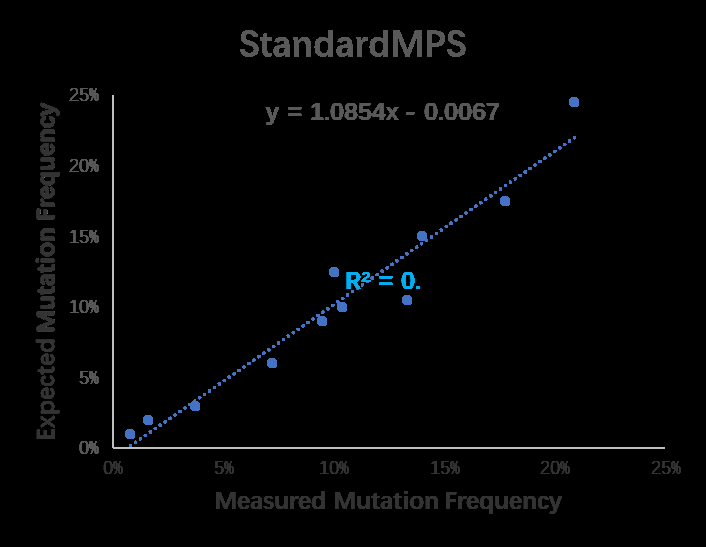
图10-1 StandardMPS与理论的突变检测频率的的相关性
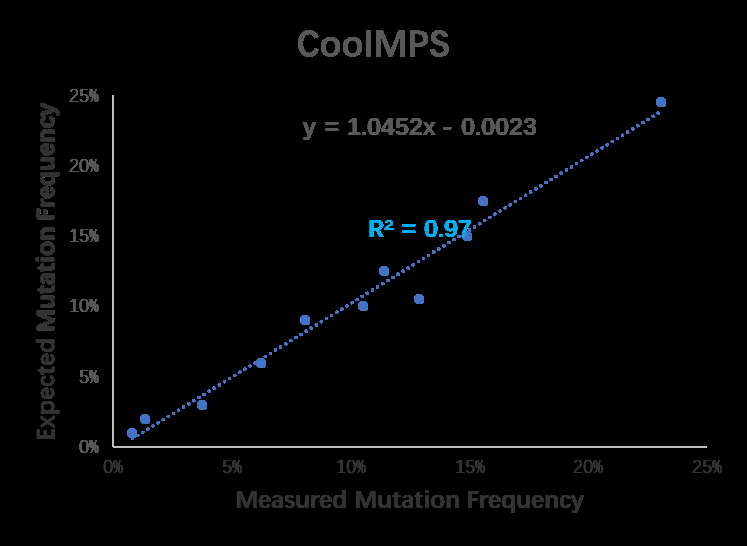
图10-2 CoolMPS与理论的突变检测频率的的相关性
-RNA-Seq:
样本:UHRR标准品
建库:MGIEasy RNA方向性文库制备试剂套装
测序平台:MGISEQ-2000(DNBSEQ-G400)
测序读长:PE100
测序试剂:CoolMPS & StandardMPS
|
|
sample |
Total Clean Bases (Gb) |
Total Genome Mapping Ratio (%) |
Total Gene Number |
Total Transcript Number |
|
CoolMPS |
Lib1 |
8.3 |
96.3 |
19446 |
31038 |
|
Lib2 |
8.8 |
96.2 |
19456 |
31154 |
|
|
Lib3 |
10 |
96.2 |
19582 |
31496 |
|
|
Lib4 |
8.5 |
96.1 |
19433 |
31108 |
|
|
StandardMPS |
Lib1 |
8.5 |
95.8 |
19495 |
31325 |
|
Lib2 |
8.4 |
95.6 |
19489 |
31180 |
|
|
Lib3 |
9 |
95.6 |
19517 |
31302 |
|
|
Lib4 |
8.8 |
95.4 |
19479 |
31244 |
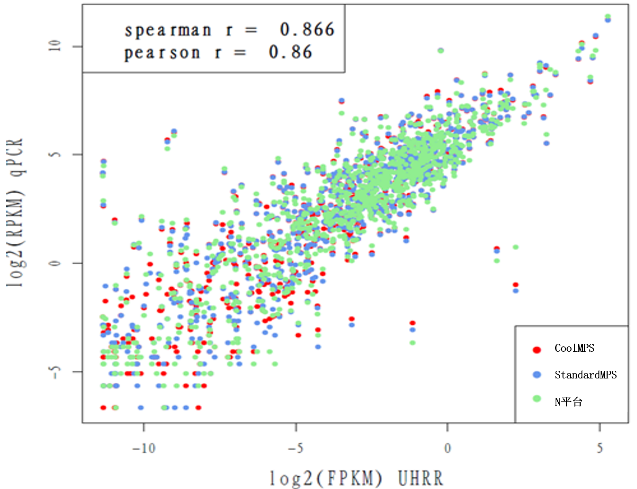
图11 CoolMPS,StandardMPS及N 平台与qPCR的相关性
CoolMPS,StandardMPS及N 平台与qPCR的相关性,Spearman r=0.866,高度一致。
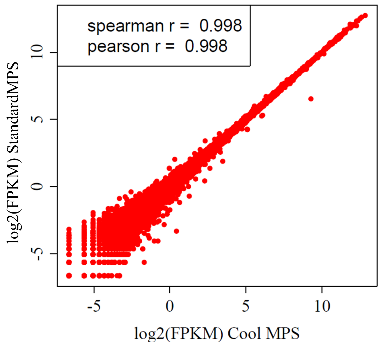
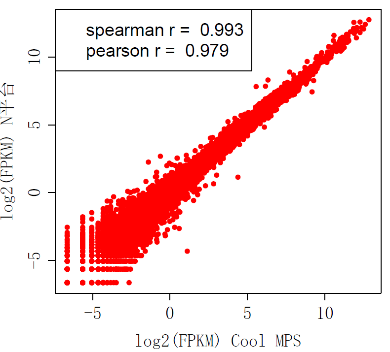
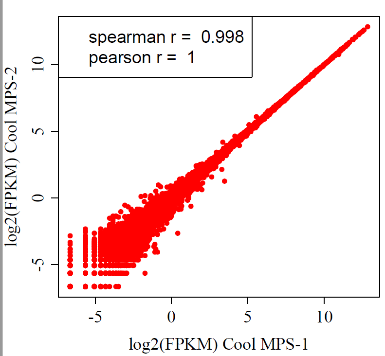
图12 CoolMPS,StandardMPS及N 平台的相关性
不同方法或相同方法测序重复性均大于0.99。
三、 CoolMPS的测序原理
大家也许对CoolMPS的测序原理依然充满好奇,它实质上可以理解为联合探针锚定聚合技术(cPAS, Combinatorial Probe-Anchor Synthesis,或称为StandardMPS)的升级版,它使用的核苷酸是碱基上未标记荧光的dNTPs(cold dNTPs)。在DNA聚合酶的作用下,cold dNTPs被聚合到测序链,然后通过和与此cold dNTPs特异结合的荧光标记的抗体(图13)来实现碱基识别。再生后,抗体从测序链上掉落,测序链上新加入的碱基完全是天然的碱基,没有任何修饰,如此循环往复,完成对DNA的测序(图14)。
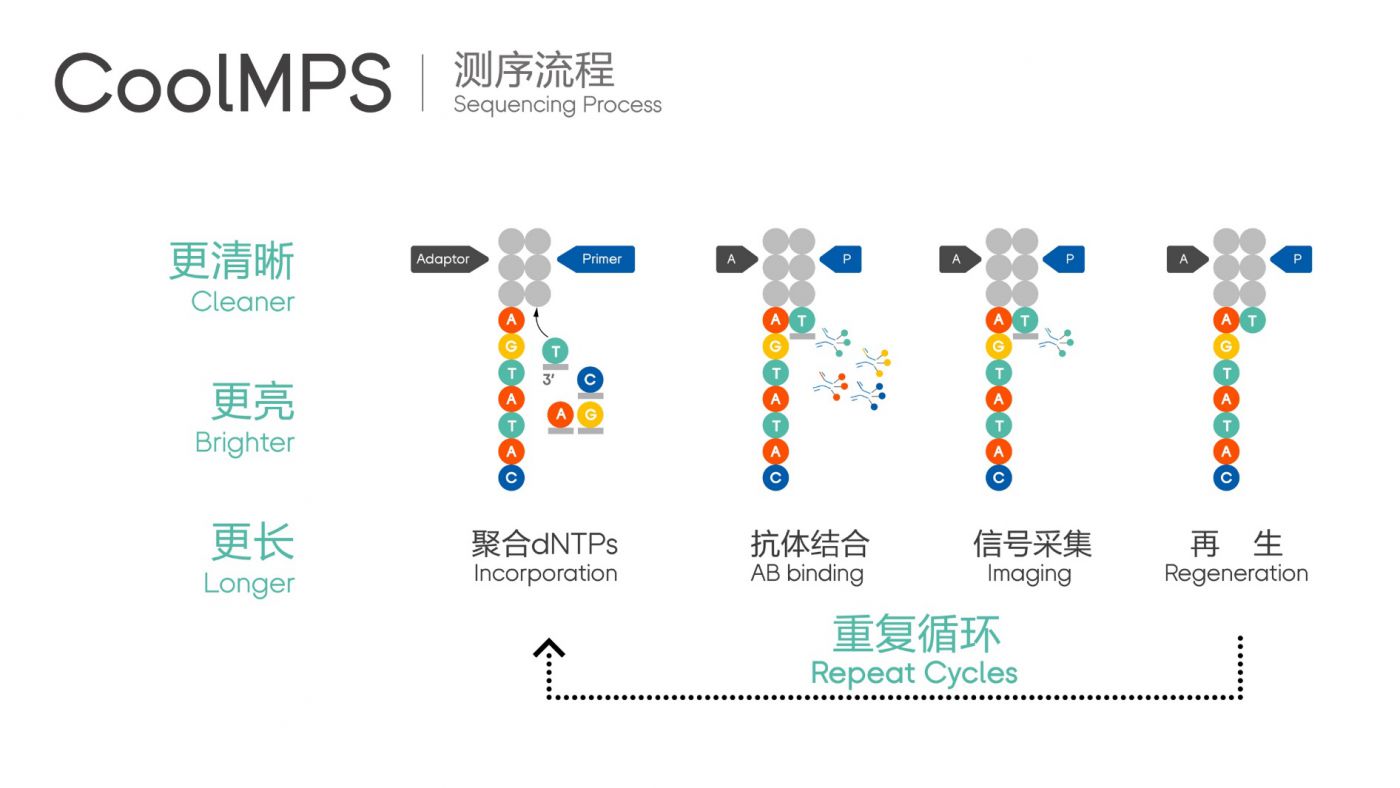
图13 CoolMPS测序原理
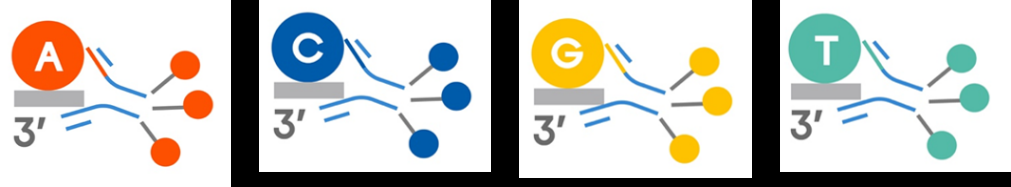
图14 CoolMPS测序时,抗体分子与碱基的结合
拥有如此多优点的CoolMPS 高通量测序试剂套装将于今年圣诞节12月25日正式开售,欢迎您一同探索它的奇妙与不凡!

 基因测序仪业务
基因测序仪业务









 技术解读
技术解读 热门应用
热门应用 组学研究(科研方向)
组学研究(科研方向) 组学研究(临床方向)
组学研究(临床方向) 文档下载
文档下载 已发表文章
已发表文章 用户成功案例
用户成功案例 在线数据集
在线数据集 多功能中心
多功能中心 客户中心
客户中心 在线支持
在线支持